Krzysztof Krawiec
New
Cel
Wygrać konkurs Konkurs ADHD-200, takze http://fcon_1000.projects.nitrc.org/indi/adhd200/index.html, czyli:
- Odnalezienie cech różnicujących mózgi osób chorych na ADHD w obrazowaniu fMRI
- Docelowo: stworzenie klasyfikatora wspomagającego diagnozowanie ADHD
- Timeline:
- Dobry prototyp kompletnego systemu: 1.06.2011
- Zbiór testujący dostajemy: 1.07.2011
- Wysłanie wyników (zaklasyfikowanych przypadków testowych) na konkurs: do 1.08.2011
Dane wejściowe
- 285 pacjentów (dzieci) z ADHD, 491 zdrowych
- 3D obraz strukturalny T1-zależny SPGR/MPRAGE voxel 1x1x1.2mm, rozdzielczość 256x254x(160..180)
- 4D obraz czynnościowy resting state BOLD (blood oxidation-level dependent) 3x3x4 mm; epi2d; TR (sampling frequency)=3000ms; ~120 próbek => ok. 6 minut zapisu
- dane kliniczne: Attach:adhd_clinical.xls (Uwaga! to jeszcze nie jest wersja ostateczna),
- Płeć (female-0, male-1)
- Wiek (w latach)
- Ręczność - praworeczny = 1, leworeczny = 0, dla osrodka 5 ujemne wartosci to leworeczny, dodatnie to praworeczny - wszyscy sa praworeczni)
- Diagnoza (ADHD i podtypy oraz norma)
- healthy control - 0
- ADHD Combined - 1
- ADHD Hyperactive - 2
- ADHD Inattentive - 3
- współczynnik inteligencji (IQ) globalny
- opisy juz mam, ale musze je przeanalizowac
- współczynnik inteligencji (IQ) werbalny
- współczynnik inteligencji (IQ) pozawerbalny
Uwagi:
- obie modalności reprezentują wartości względne
- niezbędna jest korejestracja przestrzenna pomiędzy nimi
- docelowy klasyfikator nie powinien korzystać z danych klinicznych; ale możemy je wykorzystać pomocniczo np. do uczenia klasyfikatorów
Oczekiwane wyjście:
- przypisanie pacjenta do jednej z 4 kategorii (0 - bez zaburzeń, 1..3 - różne warianty ADHD),
Wizja dataflow
A. Wstepne przetwarzanie danych
1. Segmentacja mózgu (skullstripping)
2 Segmentacja FAST do 3 kategorii strona techniczna FAST
- istota szara (GM),
- istota biala (WM),
- płyn mózgowo-rdzeniowy (CSF)
Dalsza analiza dotyczy wyłącznie wokseli GM, choć inne też będą wykorzystane np. do estymacji fluktuacji pola magnetycznego.
3 FLIRT to korejestracji z BOLD
3.1 Korzystamy z rejestracji liniowej z szescioma stopniami swobody
4 Korejestracja do atlasu za pomoca FLIRT i wygenerowanie zagregowanych przebiegów BOLD dla poszczególnych struktur mózgu
4.1 Alternatywnie możemy uzyć FNIRT czyli narzedzie rejestracji elastycznej
5 opcja: FIRST - segmentacja struktur podkorowych, na bazie atlasu;
Zdjecie tablicy: 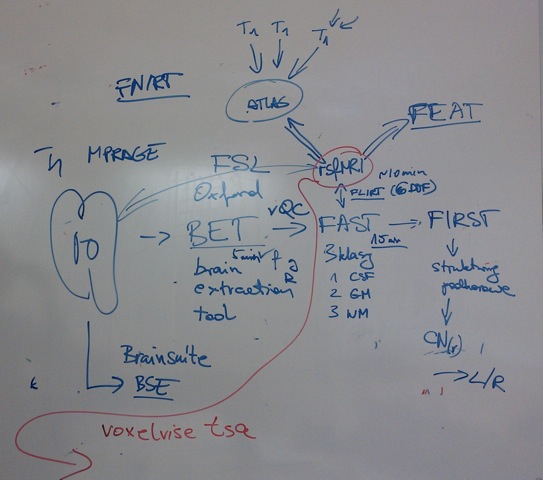
Konieczność odszumienia spektrum przebiegu czasowego obrazu ze względu na obecność:
- zmian pola magnetycznego ziemi: globalne zmiany sygnalu
- efektywności magnetycznej - na podstawie plynu mozgowo-rdzeniowego
- podatności magnetycznej - na podstawie sygnału z istoty bialej (w jaki sposob zmiany p. magnetycznego wplywaja na cala tkanke (istota biala jest slabo ukrwiona))
- korekta ruchu pacjenta.
B. Ekstrakcja cech
Cel: stworzenie (w miarę zwartej) reprezentacji danych otrzymanych z etapu A. Na dziś zakładamy że proces ten byłby raczej nienadzorowany, czyli nie korzystałby z rozpoznań pacjentów.
Zakładane dane wejściowe: zagregowane przebiegi dla poszczególnych struktur mózgu (lub pojedynczych wokseli)
Grupy cech:
- L: Cechy lokalne, tj. budowane na każdym szeregu czasowym x_i niezależnie
- DFT: Dyskretna transformata Fouriera DFT(x_i), czyli cos zblizonego do reprezentacji spektralnej w wykonaniu pp. Szymona i MP
- Wavelets? (falki)
- Autorkorelogram (wektor wspolczynnikow korelacji dla roznych opoznien)
- P: Cechy 'pairwise', tj. budowane z par szeregów czasowych (x_i,x_j)
- Korelogram
- DFT z zespolonych szeregow czasowych, tj. szeregu x_i+j*x_j (gdzie j to jednostka urojona)
- Jakas agregacja DFT(x_i) i DFT(x_j)
- G: Cechy globalne, liczone z wszystkich przebiegow czasowych (lub ich podzbioru)
- PCA
- Nonlinear PCA
- Trajektorie PCA (do doprecyzowania)
C. Klasyfikacja
Cel: Przygotowanie i nauczenie klasyfikatorów diagnozujących pacjentów na podstawie reprezentacji (jednej lub wielu) otrzymanych z etapu B
- Przewidujemy wykorzystanie klasyfikatorów złożonych
Przypisanie zadań do zespołów
(dla uproszczenia pozwalam sobie adresowac Was po imieniu)
- Szymon, Kognitywistyka 4 rok
- Część A; uruchomienie obliczeń A
- Michał,Przemko,Wojtek, Informatyka 3 rok
- 27.04: Zapoznac sie ze skrytpem pythonowym (+ plik konfiguracyjny) ktory mozna by wykorzystac do rownoleglego odpalania obliczen na maszynach w lab 1.6.21: Attach:jobrunner.zip
- Zadanie: Zapoznac sie z formatem pliku w ktorym bedziemy dostawac szeregi czasowe. Format jest prosty: plik tekstowy, kazda kolumna odpowiada pojedynczemu voxelowi; pierwsze trzy wiersze definiuja wspolrzedne (x,y,z) voxela; kolejne wiersze to szereg czasowy. Przykladowy plik w takim formacie dla pewnego pacjenta: Attach:timeSeries.zip
- Jarek, Mateusz, Informatyka 4 rok
- Zadanie: napisac funkcje w C/C++ _efektywnie_ (zadanie w całości zakończone):
- wczytujacą pliki (~50MB) i zaprojektowac strukture danych do jej przechowywania :
- konwertującą pliki .txt do .csv
- łączącą wiele plików w jeden, przekształcając każdy plik wejściowy w jedną linijkę pliku wyjściowego
- Zadanie: napisac funkcje obliczającą cehy (zadanie w większości zakończone):
- PCA
- cross-korelację
- Zadanie: implementacja Dataflow - Klasyfikacja (zadanie do zrobienia)
- Zadanie: napisac funkcje w C/C++ _efektywnie_ (zadanie w całości zakończone):
- Tomasz, Krzysztof, Informatyka 4 rok
- Zadanie: napisac funkcje implementujace cechy (zobacz wykaz cech powyzej):
- L.DFT, tj. dyskretną transformatę Fouriera z każdego szeregu z powyzszej strukutry.
- L.Autokorelacja, tj. obliczająca autokorelację dla każdego z przebiegów z osobna (dla zadanego zakresu opóźnień [1, k]
- P.Autokorelacja, tj. obliczająca korelogram dla zadanej pary przebiegów (dla zadanego zakresu opóźnień [1, k]
- Zadanie: implementacja Dataflow - Preprocessing
- Zadanie: napisac funkcje implementujace cechy (zobacz wykaz cech powyzej):
Wspólne zadania dla wszystkich
Michał Przemko Wojtek Jarek Mateusz Tomasz Krzysztof:
- zapoznanie się z formatem NIFTI http://nifti.nimh.nih.gov/
- wybadanie czy da się skorzystać z biblioteki wczytującej obrazy nifti do biblioteki OpenCV Attach:niftyOpenCV.zip Attach:niftyOpenCV.pdf
- zapoznać się z wybranymi metodami ekstrakcji cech, pod kątem (potencjalnej) analizy danych strukturalnych (T1); źródło: materiały z PiRO: Attach:metodyOpisuObrazu.pdf
- [KK] Zapoznać się z artykułem Eckera et al. Attach:adhd-svm.pdf Metoda wykorzystuje cechy strukturalne wyekstrahowane z obrazow T1-zaleznych. Cechy te opisują na różne sposoby stopień zakrzywienia/pofałdowania kory mózgowej oraz przestrzenny rozkład jej grubości. Cechy zbierane sa dla kazdego voxela grey matter osobno (choc niestety nie doczytalem sie ile w sumie jest voxeli). Klasyfikator to SVM (a nie mówiłem!). Praca zawiera nie tylko wyniki testow klasyfikacyjnych leavingj-one-out, ale takze ciekawe wizualizacje pokazujace ktore struktury korowe najlepiej odrozniaja chorych od grupy kontrolnej.
Pewna nietypowość tej pracy polega na tym że (jeżeli dobrze rozumiem), pacjenci z ADHD są tu dodatkowa grupą kontrolną, bo praca dotyczy autyzmu (ASD). Czyli sa tam 3 grupy: ASD, Control, ADHD. Ale na samą metodykę analizy danych nie ma to wpływu.
Oczywiscie na pewno nie pojdziemy dokladnie ta droga, ale z tej pracy wiele mozna sie nauczyc.
Konkretne konkluzje dla naszych prac:- rozwazyc wykorzstanie podobnych cech strukturalnych,
- w analizie obrazu bazowac na cechach typu gradient przestrzenny, ktore pozwalaja sie nam uwolnic od absolutnych jasnosci voxeli, ktore sa przeciez nieporownywalne pomiedzy pacjetami,
- rozwazyc konstruowanie osobnych klasyfikatorow dla lewej i prawej polkuli,
Otwarte kwestie/pytania:
(prosze nie usuwac pytan tylko dopisywac odpowiedzi)
- Czy rozrozniamy stopnie zaawansowania ADHD, czy tez wrzucamy wszystkich chorych do jednego worka?
- A jeżeli to drugie, to czy możemy potraktować zmienną decyzyjną w sposób porządkowy? (tj. czy decyzja '3' to cięższa odmiana ADHD niż decyzja '2'?)
- Według aktualnego mojego zrozumienia wrzucamy wszystkie podtypy do jednego worka, poniewaz w sensie klinicznym taki sens ma wspomaganie decyzji tj. chodzi o to czy mamy przeslanke zeby odroznic osobe badana od zdrowych, a nie czy mamy do czynienia z takim czy innym podtypem ADHD. Natomiast sytuacja moze sie zmienic jesli w wyniku naszych analiz okaze sie, ze grupy 1, 2, 3 sa znaczaco heterogenne i mamy metody do odroznienia tych grup od siebie (w co jak na razie watpie, ale chetnie bede sie mylic) - MP
Specyfikacja implementacji Dataflow
Preprocessing danych
Skrypt do przekształcenia surowych przebiegów czasowych w pliki zawierające cechy przeznaczone do klasyfikacji:
- język: Python
- zdalna instalacja na jednostkach roboczych (węzły robocze instalowane są zdalnie przez ssh, zgodnie z listą konfiguracyjną)
- zachowywanie wyników pośrednich; ponowne wyliczanie w przypadku zmiany plików zależnych bądź na życzenie użytkownika
- zawartość pliku określana na podstawie nazwy ścieżki do pliku oraz jego nazwy; dane zawarte w ścieżce (katalogi):
- nazwa ośrodka
- id pacjenta
dane zawarte w nazwie pliku:
- typ danych (np. przebieg czasowy, przetworzony przebieg czasowy, etc.)
- skrypt składa się z następujących etapów (części):
- uruchomienie dowolnego skryptu, który zostanie wykonany dla wszystkich pacjentów (struktura danych jest identyczna w katalogu każdego pacjenta), w tym uruchomienie zadań związanych z analizą szeregów czasowych dla pojedynczego mózgu (np. FFT, autokorelacja, cross-koeralcja pomiędzy strukturami)
- każdy krok obliczeń dla poszczególnych pacjentów, jak również lista ścieżek folderów, zawierających foldery pacjentów, zawarty jest w pliku konfiguracyjnym zawierający listę poleceń, które należy wykonać (w postaci programów / skryptów do wykonania). Zakładamy przy tym identyczną strukturę katalogów pacjentów. Wszystkie podane w argumentach ścieżki są względne (poza ścieżką do programu), ich katalogiem głównym jest katalog danego pacjenta.
- agregacja otrzymanych danych na węźle głównym (rozwijanie pojedynczych plików w jedną linię i scalenie linii w jeden plik), agregacja jest funkcją Dataflow i przebiega zawsze w następujący sposób: podawana jest względna ścieżka do pliku, w której każdy z pacjentów ma jakąś cechę (przyjmujemy, że dana cecha w każdym katalogu z id pacjenta jest w tym samym pliku / ścieżce). Funkcja przechodzi po wszystkich katalogach pacjentów i zapisuje id pacjenta jako pierwszą krotkę wiersza, a jako kolejne oddzielone spacjami wartości atrybutów. W wyniku działania agregacji otrzymuje się plik, w którym każdy pacjent opisany jest wierszem cech, pierwsza z nich to jego id, a kolejne są dowolne. Zakłada się możliwość opisania pacjenta dowolną liczbą cech, aczkolwiek dla każdego pacjenta musi być ona identyczna. UWAGA: Zgodnie z założeniami pliki zawierające poszczególne cechy mają strukturę wektora lub macierzy (kolumny oddzielone są spacjami a wiersze znakami nowej linii). Funkcja agregująca musi umożliwić określenie marginesów (możliwość pominięcia pewnych wierszy i kolumn, w szczególności początkowych - ponieważ mogą to być nieistotne dane takie jak nagłówki lub współrzędne wokseli / nazwy struktur), jeżeli plik jest macierzą, wówczas musi zostać on przepisany jako wektor wiersz po wierszu.
- DEPRECATED (nie ma sensu automatyzować zadań na tak wysokim poziomie abstrakcji, są osobne programy do PCA i transformacji plików, w szczególności użycie RapidMinera z GUI do tworzenia plików WEKI):
- uruchomienie zadań działających na zagregowanych danych (np. PCA, niezalecane ze względu na chęć zachowania wszystkich przekształceń przestrzeni atrybutów w modelu klasyfikatora, choć może okazać się konieczne w przypadku plików o bardzo dużym rozmiarze)
- transformacja zagregowanych danych (wszystkich) do formatu CSV, wzbogacenie informacji o klasę decyzyjną
- uruchomienie modułu klasyfikującego
Klasyfikacja:
- implementacja w języku Java z wykorzystaniem pakietu Weka
- implementacja lub wykorzystanie filtrów / operatorów selekcji, konwersji (w tym przekształcenia przestrzeni), zmiany typów atrybutów
- implementacja lub wykorzystanie klasyfikatorów złożonych, w szczególności:
- Random Subspace (SVM)
- Random spherical oracle (SVM)
- Random linear oracle (SVM)
- możliwość stworzenia przepływu w postaci:
- selekcja / konwersja atrybutów (R2, PCA, etc., jeżeli operacja nie jest związana z klasami - umożliwienie wykonania jej dla dwóch zbiorów danych - etykietowanych i nieetykietowanych)
- konwersja typów atrybutów (pomiędzy wartościami: numerycznymi, nominalnymi oraz binominalnymi)
- stworzenie modelu (instancji klasyfikatora)
- testowanie klasyfikatora (klasa opakowująca klasyfikator, testująca skuteczność jego działania, nie wpływająca w żaden sposób na proces uczenia)
- możliwość zapisania schematu całego przepływu w postaci pliku konfiguracyjnego, w którym zapisane są kolejno
- sekcja z listą zmiennych wraz z zakresami i krokiem modyfikowania lub listą kolejnych wartości
- ścieżka do pliku ze zbiorem treningowym
- ścieżka do pliku z modelem (konieczne użycie wszystkich zadeklarowanych zmiennych w nazwie)
- ścieżka do pliku ze zbiorem do klasyfikacji (opcjonalnie)
- sekcja z listą, linijka pod linijką, przekształceń zbioru atrybutów (każda linijka zawiera nazwę klasy z przekształceniem oraz parametry, parametry te są inne dla każdego przekształcenie, jako wartości parametrów mogą być użyte zmienne)
- klasyfikator wraz z parametrami (możliwe użycie zmiennych jako wartości parametrów)
- sposób testowania klasyfikatora (możliwe wartości: k-fold-CV, split-x, gdzie k jest liczbą naturalną > 1, a x liczbą z pomiędzy 0 a 1 odpowiadająca części zbioru wejściowego, która będzie uważana za zbiór uczący)
- seed dla klasy random
- możliwość zapisania modelu przepływu do pliku binarnego (w celu późniejszej klasyfikacji nowych rekordów):
- plik zawiera model (operacje na atrybutach, nauczony klasyfikator)
- plik zawiera wszystkie informacje zawarte w pliku konfiguracyjnym
- jeżeli wykonano testy na trafność klasyfikacji plik posiada dodatkowo informacje na temat ich wyników (mierzenie precision i recall dla wszystkich klas, trafności całej klasyfikacji w postaci średnich oraz, w przypadku testowania z użyciem cross-validation, odchyleń standardowych)
- jeżeli plik konfiguracyjny zawiera klasyfikator ze zmiennymi parametrami, wówczas dla każdego klasyfikatora tworzony jest osobny plik zawierający przepływ
- zpisanie pliku CSV o identycznej nazwie jak plik z modelem, zawierającego linijkę z nazwą pliku w pierwszym polu oraz wartości statystyk związanych z trafnością klasyfikacji jako kolejne pola, pola oddzielone są średnikami (zapis taki umożliwi późniejszą agregację wszystkich przeprowadzonych testów)
- uruchomienie programu z jednym argumentem w postaci pliku konfiguracyjnego lub katalogu z plikami (program sam rozpozna co podano, brak dodatkowych przełączników)
- stworzenie dodatkowej klasy aplikującej model do pliku, który ma zostać zaklasyfikowany
- wszędzie gdzie jest to możliwe używany będzie obiekt klasy Random o ustalonej wartości seed w celu zapewnienia powtarzalności tych samych eksperymentów
Komentarze do implementacji Dataglow:
- jeżeli pewna funkcjonalność opisana w wymaganiach istnieje w Wece, wówczas jest ona wykorzystywana (np. bagging przy tworzeniu klasyfikatorów złożonych)
- konwersja typów atrybutów może być przydatna dla wyszukiwania wzorców sekwencji
- PCA będzie raczej uruchamiane poprzez moduł klasyfikujący, aby zachować spójność (wszelkie inne przekształcenia lub ograniczenia przestrzeni atrybutów mają miejsce w module klasyfikującym, ponadto w łatwy sposób można zapisać wówczas cały model / workflow, któremu poddane zostały dane, aby móc stworzyć klasyfikację dla nowych danych)
- korzystne byłoby, po otrzymaniu nowych danych, policzyć PCA dla wszystkich mózgów (łącznie z tymi, które mają zostać poddane klasyfikacji), dlatego zostanie dodana możliwość policzenia PCA dla dwóch plików wejściowych
- klasyfikatory oraz inne operacje zapisane są w pliku konfiguracyjnym jako nazwy klas, dzięki temu poprzez użycie mechanizmu Reflection zostaną wczytane do modułu jako odpowiednie klasy
- plik z modelem zawiera informacje z pliku konfiguracyjnego, aby w przypadku usunięcia pliku konfiguracyjnego znany był dokładny przebieg powstania klasyfikatora
- w konfiguracji w module klasyfikatora ustawienia związane z preprocessingiem zapisane są jedynie w nazwie pliku wejściowego
Literatura:
- Soliva JC, Fauquet J, Bielsa A, Rovira M, Carmona S, Ramos-Quiroga JA, Hilferty J, Bulbena A, Casas M, Vilarroya O. Quantitative MR analysis of caudate abnormalities in pediatric ADHD: proposal for a diagnostic test. Psychiatry Res. 2010 Jun 30;182(3):238-43. przykładowy artykuł opisujący wykorzystanie cech jądra ogoniastego do diagnozowania ADHD Attach:ogoniaste.pdf
- Aberrant temporal and spatial brain activity during rest in patients with chronic pain. Malinen S, Vartiainen N, Hlushchuk Y, Koskinen M, Ramkumar P, Forss N, Kalso E, Hari R. Proc Natl Acad Sci U S A. 2010 Apr 6;107(14):6493-7. Zastosowanie analizy spektrum sygnału BOLD do identyfikacji pacjentow z przewlekłym bólem
- Dosenbach, N.U.F., Nardos, B., Cohen, A. L., Fair, D. A., Power, J. D., Church J.A., Nelson, S. M., Wig G.S., Vogel, A.C., Lessov-Schlaggar, C.N., Barnes, K.A., Dubis, J.W., Feczko, E., Coalson, R. S., Pruett Jr.,J.R., Barch, D. M., Petersen, S. E., Schlaggar, B. L. (2010). Prediction of Individual Brain Maturity Using fMRI. Science 329, 1358-1361. Attach:Dosenbach2010.pdf
- Suplement do powyzszego artykulu. Attach:Dosenbach_sup2010.pdf
Klasyfikator
- KK: Prawdopodobnie w pierwszym podejsciu uzyjemy liniowego klasyfikatora SVM. Metody weryfikacji istotnosci wyniku: walidacja krzyżowa i permutation testing.
- Zhu CZ, Zang YF, Cao QJ, Yan CG, He Y, Jiang TZ, Sui MQ, Wang YF (2008), Fisher discriminative analysis of resting-state brain function for attention-deficit/hyperactivity disorder. Neuroimage 40:110-120. Attach:Zhu2008.pdf
- Yufeng Zang, Tianzi Jiang, Yingli Lu, Yong He, and Lixia Tian (2004) .Regional homogeneity approach to fMRI data analysis. Neuroimage 22: 394-400. Attach:Zang2004.pdf
- Nicole Lazar, The Statistical Analysis of Functional MRI Data (2008) ISBN: 978-0-387-78190-7 Attach:Lazar.pdf
- M. Lopez, J. Ramirez, J.M. Gorriz et al., Automatic System for Alzheimer’s Disease Diagnosis Using Eigenbrains and Bayesian Classification Rules, Springer-Verlag Berlin Heidelberg 2009 Attach:eigenbrains_bayes.pdf
- I. Alvarez, J.M. Gorriz, J. Ramirez et al., Alzheimer’s Diagnosis Using Eigenbrains and Support Vector Machines, Springer-Verlag Berlin Heidelberg 2009 Attach:eigenbrains_svm.pdf