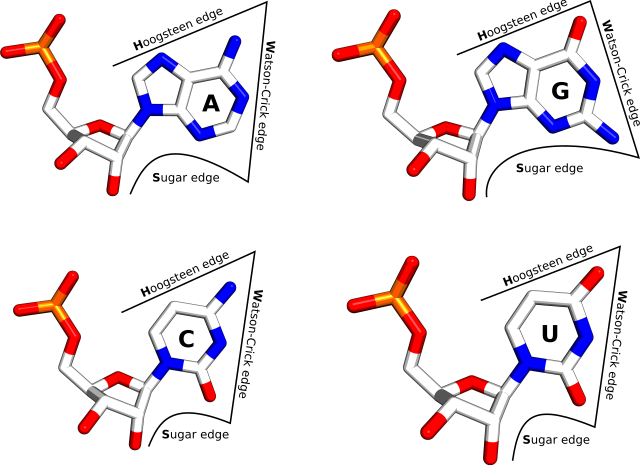
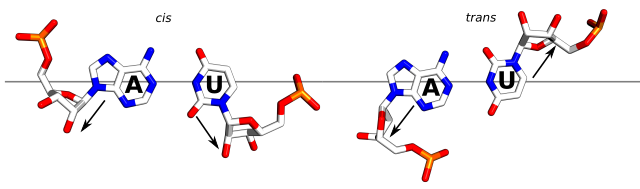


Pewne kombinacje sekwencji i par niekanonicznych powtarzają się w wielu strukturach
Przyjmują one często charakterystyczny kształt 3D wynikający z kombinacji par niekanonicznych
Lista najbardziej znanych modułów / motywów 3D (na podstawie (Ge2018?), grafika 2D wykonana przy pomocy VARNA (Darty2009?)):
| Nazwa | Wizualizacja | Struktura 2D |
|---|---|---|
| GNRA | 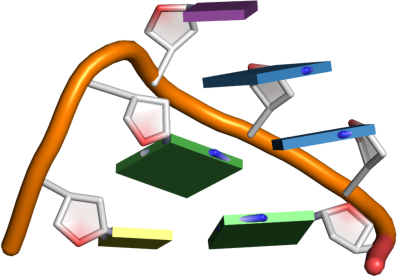 |
|
| UNCG | 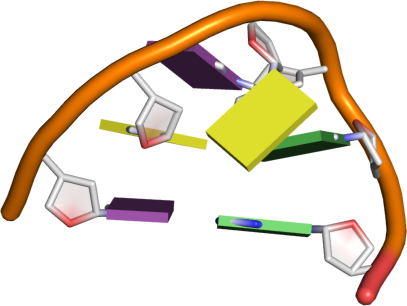 |
|
| T-loop | 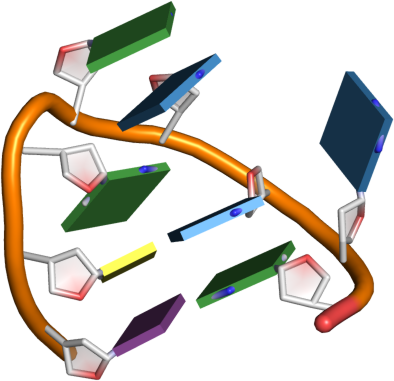 |
|
| Sarcin-ricin |  |
|
| Kink-turn |  |
|
| Hook-turn | 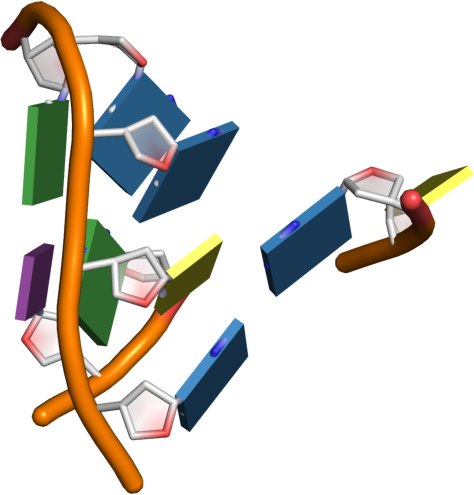 |
|
| C-loop |  |
|
| E-loop | 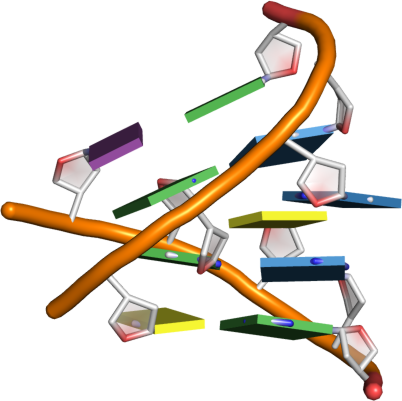 |
Napisz program do wyszukiwania motywu:
Motyw powinien być odnaleziony wyłącznie na podstawie wzorca parowań niekanonicznych (a nie na podstawie sekwencji):
GNRA:
cWW (....)
tSH .(..).UNCG:
cWW (....)
tSW .(..).T-loop:
cWW (......)
tSH .(...)..E-loop:
cWW (...( & )...)
tSH .(... & ...).
tWW ..(.. & ..)..
tHS ...(. & .)...C-loop:
cWW (...(( & ).)...)
tWH ..(... & ....)..
tHS ...(.. & ...)...Dane do analizy zostały wygenerowane przez narzędzie DSSR i zapisane są w formacie JSON
Najważniejsze informacje znajdują się w tablicy
pairs i nts:
"pairs": [
{
"index": 1,
"nt1": "AA.G9",
"nt2": "AA.C25",
"bp": "G-C",
"name": "WC",
"Saenger": "19-XIX",
"LW": "cWW",
"DSSR": "cW-W"
},
...
],
"nts": [
{
"index": 1,
"index_chain": 1,
"chain_name": "AA",
"nt_resnum": 4,
"nt_name": "U",
"nt_code": "U",
"nt_id": "AA.U4",
...
},
...
]Z tablicy pairs odczytać można klasyfikację pary w
notacji Leontisa-Westhofa (pole LW) oraz identyfikatory
nukleotydów (pola nt1 i nt2)
Z tablicy nts odczytać można indeks, nazwę łańcucha,
identyfikator oraz jednoliterową nazwę (pola index,
chain_name, nt_id,
nt_code):
Przykład:
Fragment pliku JSON:
"pairs": [
{
"nt1": "A.C11",
"nt2": "A.G16",
"LW": "cWW"
},
{
"nt1": "A.U12",
"nt2": "A.G15",
"LW": "tSW"
},
...
],
"nts": [
{
"index": 11,
"chain_name": "A",
"nt_code": "C",
"nt_id": "A.C11"
},
{
"index": 12,
"chain_name": "A",
"nt_code": "U",
"nt_id": "A.U12"
},
{
"index": 13,
"chain_name": "A",
"nt_code": "U",
"nt_id": "A.U13"
},
{
"index": 14,
"chain_name": "A",
"nt_code": "C",
"nt_id": "A.C14"
},
{
"index": 15,
"chain_name": "A",
"nt_code": "G",
"nt_id": "A.G15"
},
{
"index": 16,
"chain_name": "A",
"nt_code": "G",
"nt_id": "A.G16"
},
...
]Mamy zatem parę (11,16) typu cWW i (12,15) typu tSW
Różnica indeksów w parze cWW wynosi 5
Nukleotydy od 11 do 16 należą do tego samego łańcucha
A zatem mamy motyw UNCG: 11 CUUCGG 16
Napisany program powinien odczytać podany plik JSON, znaleźć w
nim odpowiedni motyw i wypisać w postaci
indeks-5' sekwencja indeks-3'
W przypadku E-loop i C-loop motyw składa się z dwóch strandów (potencjalnie z różnych łańcuchów), więc trzeba wypisać dwie informacje w takiej postaci