Wstęp
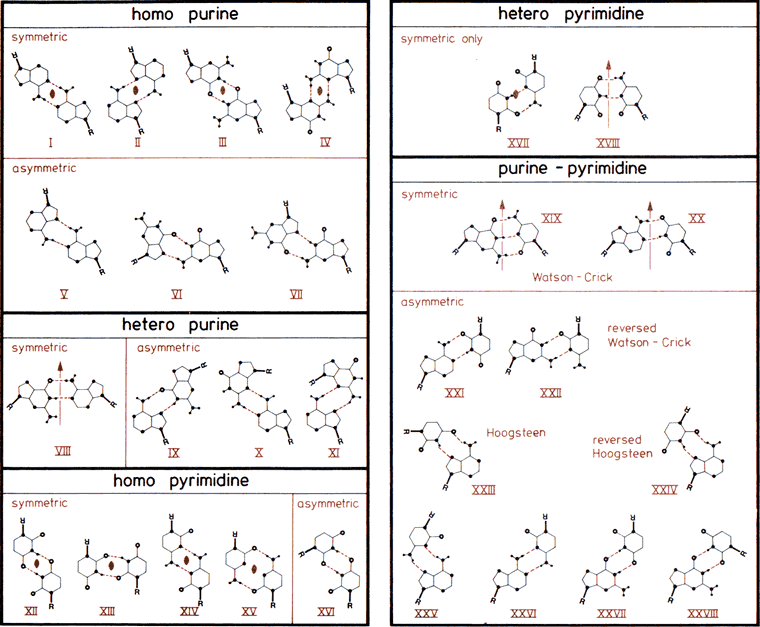
Kody Saengera
- Pierwszą powszechnie stosowaną klasyfikację par zasad zaproponował Wolfram Saenger
- W notacji Saengera, określone konfiguracje par zasad otrzymały kolejne numery (zapisywane przy użyciu cyfr rzymskich)
- Kanoniczne pary Watsona-Cricka mają numery XIX oraz XX
- Oryginalna tabela kodów Sangera:
Notacja Leontisa-Westhofa
- Dalsze badania nad strukturą RNA wykazały jednak większą zmienność i elastyczność niż to było zapisane w 28 konfiguracjach Saengera
- Poza tym, kody Saengera są po prostu kolejnymi numerami, które same w sobie nie niosą żadnej informacji (np. nie można powiedzieć, żeby między kodem I a II była "taka sama różnica" jak między IX a X)
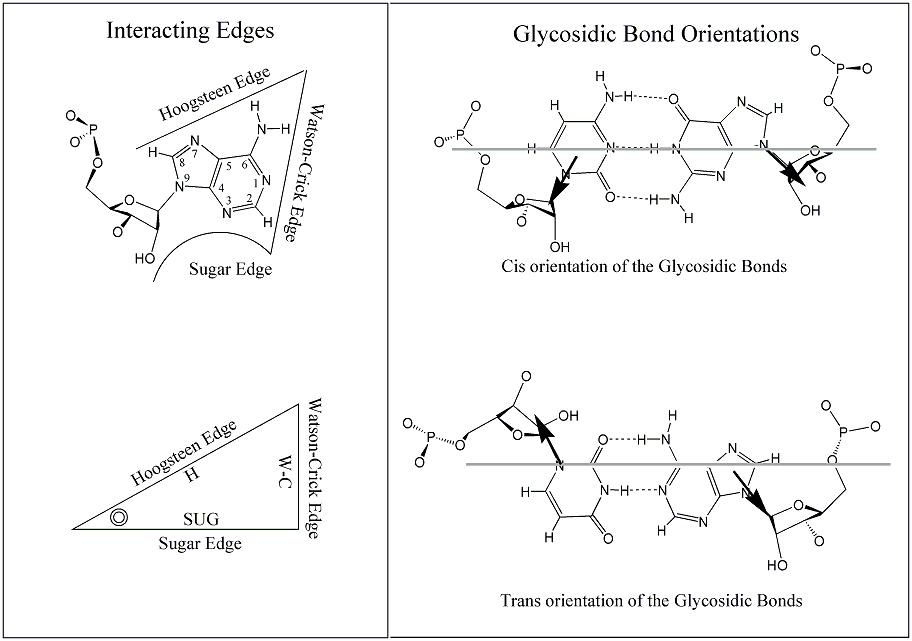
- Leontis i Westhof w pracy "Geometric nomenclature and classification of RNA base pairs" zaproponowali nową, ogólniejszą notację:
- W każdym nukleotydzie wyznaczyli oni trzy krawędzie
- Każdą parę da się zaklasyfikować albo jako cis albo jako trans w zależności od umiejscowienia rybozy względem zasad biorących udział w parowaniu
- Ideę tę pokazuje następujący schemat:
- W ogólności dla każdej z reszt tworzących parę, może uczestniczyć w tym dowolna krawędź i każda z takich par może być albo cis albo trans, co daje łącznie zestaw 12 konfiguracji:
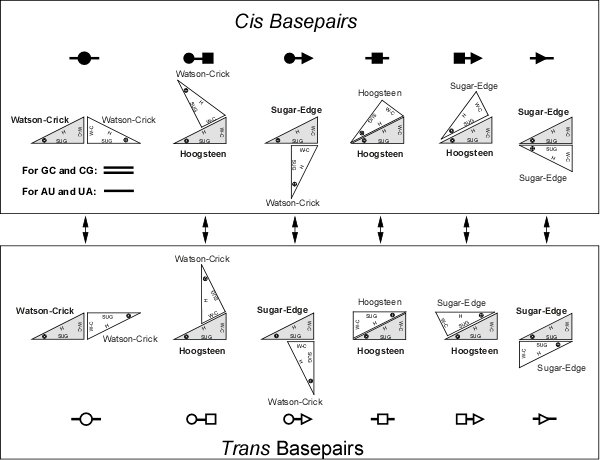
- Para w notacji Leontisa-Westhofa jest zatem oznaczona najczęściej trzyliterowym skrótem np. cWW (czyli cis, Watson-Crick, Watson-Crick) albo tHS (czyli trans, Hoogsteen, Sugar)
- Dzięki temu już w samym zaklasyfikowaniu pary odnotowuje się to jaka ta para jest
- Kodów w notacji Leontisa-Westhofa jest mniej niż Saengera, więc nie jest on tak ściśle jednoznaczny (np. kod cWW może oznaczać kanoniczne wiązanie Watsona-Cricka G-C lub A-U, ale też inne konfiguracje). Z drugiej strony, znamy dziś mnóstwo rodzajów parowań niemieszczących się w zamkniętej tabeli kodów Saengera, natomiast doskonale opisanych przy pomocy krawędzi oraz informacji o cis/trans
- Tabelę z odpowiednikami kodów w obu notacjach można znależć tutaj
Pary wielokrotne
- W wielkich i złożonych cząsteczkach RNA mogą formować się tryplety lub nawet jeszcze bardziej liczne grupy reszt tworzących wzajemną sieć wiązań wodorowych:
- triplet:
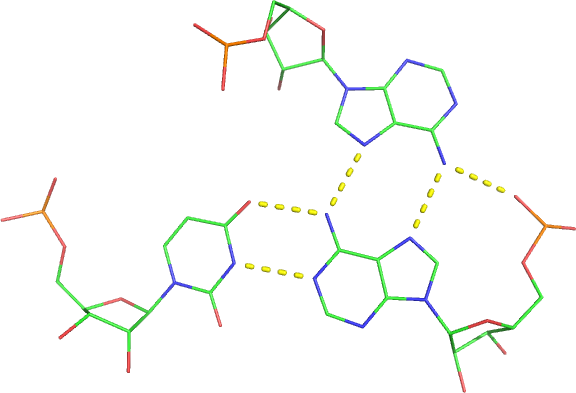
- quadruplet:

- quintuplet:
- triplet:
- Trójki zasad i ich różne konfiguracje można podejrzeć tutaj
Zbiorcze zestawienie
- Warto przyjrzeć się danym zebranym w bazie danych BPS
- Na stronie Atlas / Base pair można sprawdzić jakie są różne konfiguracje parowań określonych zasad azotowych, jak często one występują i jakie są ich parametry
- Na stronie Atlas / Higher order z kolei zobaczyć można podobny opis dla multipletów
Formaty danych
- Najprostszym formatem do opisu par zasad jest BPSEQ:
1 A 0
2 C 4
3 U 0
4 G 2- Kolumna 1: numer reszty
- Kolumna 2: rodzaj reszty
- Kolumna 3: numer sparowanej reszty (0 = brak pary)
- Istotną alternatywą jest format dot-bracket, w którym posługujemy się kropkami i nawiasami: (otwierający i zamykający nawias to para w strukturze RNA)
.brak pary()podstawowe parowanie[]pseudowęzeł pierwszego rzędu{}pseudowęzeł drugiego rzędu<>pseudowęzeł trzeciego rzędu
- Przykład:
GCGGAUUUAgCUCAGuuGGGAGAGCgCCAGAcUgAAgAucUGGAGgUCcUGUGuuCGaUCCACAGAAUUCGCACCA
(((((((..((((.....[..)))).((((.........)))).....(((((..]....)))))))))))).... - W dot-bracket możemy opisać wprost pseudowęzły, a także wyraźniej widać gdzie znajdują się dłuższe sparowane fragmenty
Narzędzia do ekstrakcji par zasad ze struktur trzeciorzędowych RNA
- Istnieje kilka podejść stosowanych w najważniejszych narzędziach do analizy struktur RNA:
- RNAView: wykorzystuje reguły geometryczne (odległości między określonymi atomami)
- MC-Annotate: ocenia na podstawie prawdopodobieństwa sparowania się dwóch zadanych nukleotydów
- 3DNA/DSSR: klasyfikuje na podstawie parametrów, które dla sparowanych zasad przyjmują określone wartości. Parametry te w biochemii RNA przedstawia następujący schemat:
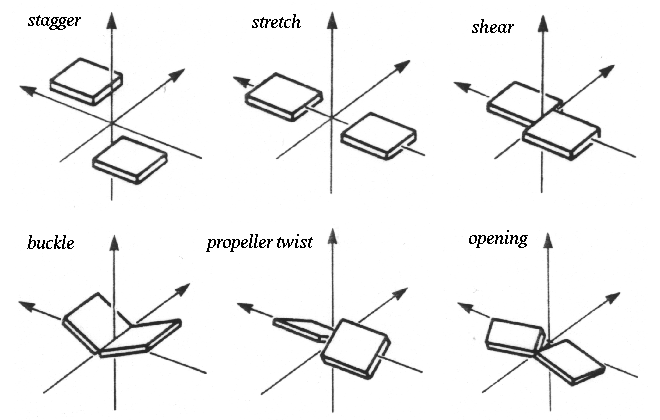
Napisz program, który dla zadanego pliku PDB zawierającego strukturę RNA odnajdzie wszystkie pary kanoniczne. Wykorzystaj dane z bazy BPS dla par:
Weź pod uwagę tylko dane z Hydrogen-bonding pattern: nazwy atomów, średnia odległość i odchylenie standardowe. Dla wejściowej cząsteczki sprawdź reszty w trybie każdy-z-każdym, dla par G-C lub A-U wyznacz odległość między wskazanymi atomami i jeśli będzie ona mniejsza lub równa średniej odległości + 3*odchylenie standardowe to uznaj parę za obecną w strukturze. Na wyjściu, wypisz strukturę drugorzędową w formacie BPSEQ.
Użyteczne linki:
Upraszczające założenia:
- Analizowane będą tylko struktury składające się z jednego modelu, jednego łańcucha i bez "insertion" tzn. każda reszta jest jednoznacznie reprezentowana przez parę (numer, nazwa)
- Analizowane będą tylko reszty niemodyfikowane tzn. o nazwach dokładnie takich: A, C, G i U
Weryfikacja Plik do weryfikacji: 1DDY_A.pdb
$ ./bpseq.py 1DDY_A.pdb
1 G 0
2 G 0
3 A 0
4 A 0
5 C 0
6 C 0
7 G 22
8 G 21
9 U 20
10 G 0
11 C 30
12 G 33
13 C 32
14 A 0
15 U 0
16 A 0
17 A 0
18 C 28
19 C 26
20 A 9
21 C 8
22 C 7
23 U 0
24 C 0
25 A 0
26 G 19
27 U 0
28 G 18
29 C 0
30 G 11
31 A 0
32 G 13
33 C 12
34 A 0
35 A 0
1 G 0
2 G 0
3 A 0
4 A 0
5 C 0
6 C 0
7 G 22
8 G 21
9 U 20
10 G 0
11 C 30
12 G 33
13 C 32
14 A 0
15 U 0
16 A 0
17 A 0
18 C 28
19 C 26
20 A 9
21 C 8
22 C 7
23 U 0
24 C 0
25 A 0
26 G 19
27 U 0
28 G 18
29 C 0
30 G 11
31 A 0
32 G 13
33 C 12
34 A 0
35 A 0
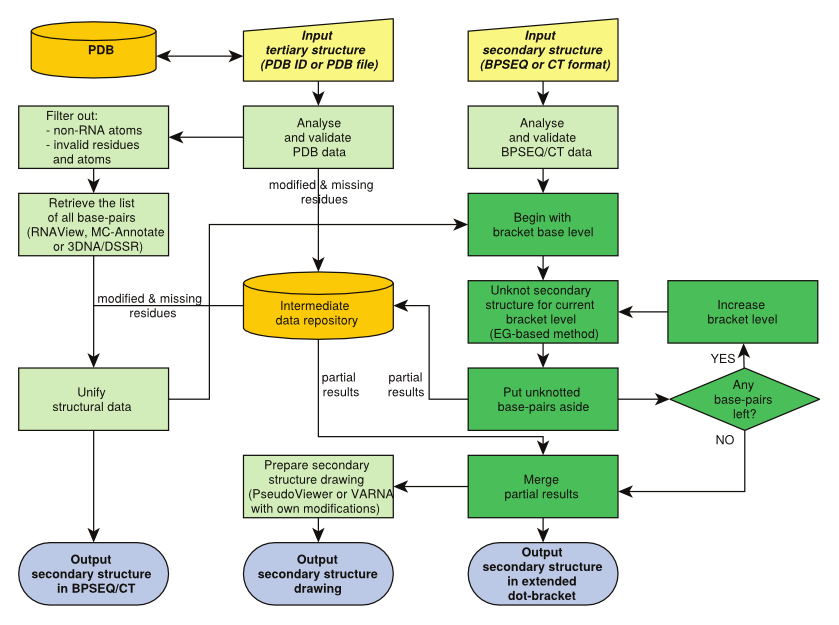
Określanie struktury drugorzędowej na podstawie modelu 3D
- Wymienione wcześniej narzędzia potrafią określić, które reszty tworzą pary, ale jeśli mamy do czynienia z pseudowęzłami (interakcjami przestrzennymi) to informacja o parowaniu nie wskazuje jednoznacznie struktury drugorzędowej
- W tym celu powstało narzędzie RNApdbee, które dla wejściowej struktury RNA wyznacza zbiór par zasad korzystając z jednego z dostępnych narzędzi, a następnie rozwiązuje problem takiego podziału zbioru, żeby wynik można było optymalnie zapisać w formacie dot-bracket (więcej szczegółów w publikacji)
- Schemat działania RNApdbee jest następujący: