Informacje sekwencyjne
- Opis formatu FASTA:
- Linia komentarza, linie z sekwencją:
>gi|5524211|gb|AAD44166.1| cytochrome b [Elephas maximus maximus]
LCLYTHIGRNIYYGSYLYSETWNTGIMLLLITMATAFMGYVLPWGQMSFWGATVITNLFSAIPYIGTNLV
EWIWGGFSVDKATLNRFFAFHFILPFTMVALAGVHLTFLHETGSNNPLGLTSDSDKIPFHPYYTIKDFLG
LLILILLLLLLALLSPDMLGDPDNHMPADPLNTPLHIKPEWYFLFAYAILRSVPNKLGGVLALFLSIVIL
GLMPFLHTSKHRSMMLRPLSQALFWTLTMDLLTLTWIGSQPVEYPYTIIGQMASILYFSIILAFLPIAGX
IENY - Symbole dla kwasów nukleinowych:
| Nucleic Acid Code | Meaning | Mnemonic |
| A | A | Adenine |
| C | C | Cytosine |
| G | G | Guanine |
| T | T | Thymine |
| U | U | Uracil |
| R | A or G | puRine |
| Y | C, T or U | pYrimidines |
| K | G, T or U | bases which are Ketones |
| M | A or C | bases with aMino groups |
| S | C or G | Strong interaction |
| W | A, T or U | Weak interaction |
| B | not A (i.e. C, G, T or U) | B comes after A |
| D | not C (i.e. A, G, T or U) | D comes after C |
| H | not G (i.e., A, C, T or U) | H comes after G |
| V | neither T nor U (i.e. A, C or G) | V comes after U |
| N | A C G T U | Nucleic acid |
| X | masked | |
| - | gap of indeterminate length |
- Symbole dla białek:
| Amino Acid Code | Meaning |
| A | Alanine |
| B | Aspartic acid or Asparagine |
| C | Cysteine |
| D | Aspartic acid |
| E | Glutamic acid |
| F | Phenylalanine |
| G | Glycine |
| H | Histidine |
| I | Isoleucine |
| J | Leucine or Isoleucine |
| K | Lysine |
| L | Leucine |
| M | Methionine |
| N | Asparagine |
| O | Pyrrolysine |
| P | Proline |
| Q | Glutamine |
| R | Arginine |
| S | Serine |
| T | Threonine |
| U | Selenocysteine |
| V | Valine |
| W | Tryptophan |
| Y | Tyrosine |
| Z | Glutamic acid or Glutamine |
| X | any |
| * | translation stop |
| - | gap of indeterminate length |
- Ensembl to zcentralizowane, europejskie źródło danych sekwencyjnych: genomy wraz z adnotacjami, wzajemne odniesienia do innych baz (np. taksonomicznych), itp.
- Amerykański odpowiednik to NCBI, a japoński to DDBJ
- Możliwości Ensembl: (źródło)
- View genes along with other annotation along the chromosome
- View alternative transcripts (including splice variants) for a gene
- Explore homologues and phylogenetic trees across more than 50 species for any gene
- Compare whole genome alignments and conserved regions across species
- View microarray sequences that match to Ensembl genes
- View ESTs, clones, mRNA and proteins for any chromosomal region
- Examine single nucleotide polymorphisms (SNPs) for a gene or chromosomal region
- View SNPs across strains (rat, mouse), populations (human), or even breeds (dog)
- View positions and sequence of mRNA and protein that align with an Ensembl gene
- Upload your own data
- Use BLAST, or BLAT, a similar sequence alignment search tool, against any Ensembl genome
- Export sequence, or create a table of gene information with BioMart
- Use the Variant Effect Predictor
- Metody porównywania sekwencji:
Following the discovery of a previously unknown gene in the mouse, a scientist will typically perform a BLAST search of the human genome to see if humans carry a similar gene
- Przejdź na stronę Ensembl
- Wybierz opcję przeglądania genomu myszy
- Wyszukaj przykładowego genu Cntnap1
- Sprawdź opcje "Gene tree" oraz "Orthologues" by wykorzystać obliczone już wcześniej dopasowania sekwencji między różnymi organizmami
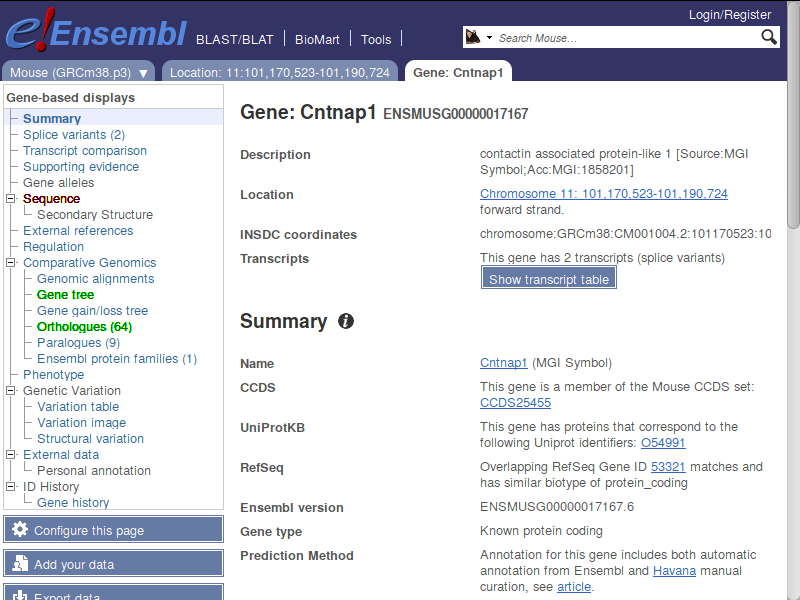
- Żeby zasymulować, że to nowo odkryty gen, wykonaj porównanie jego sekwencji z genomem człowieka:
- Kliknij w "Sequence"
- Wybierz "BLAST this sequence"
- Wybierz w "Search against" opcję "Human (Homo sapiens)" i kliknij RUN
- Sprawdź wyniki alignmentu dla kilku najlepiej dopasowanych fragmentów
Which bacterial species have a protein that is related in lineage to a certain protein with known amino-acid sequence?
- Wejściowa sekwencja aminokwasów:
MFEPMELTNDAVITVIGVGGGGGNAVEHMVRERIEGVEFFAVNTDAQALRKTAVGQTIQI
GSGITKGLGAGANPEVGRNAADEDREALRAALEGADMVFIAAGMGGGTGTGAAPVVAEVA
KDLGILTVAVVTKPFNFEGKKRMAFAEQGITELSKHVDSLITIPNDKLLKVLGRGISLLD
AFGAANDVLKGAVQGIAELITRPGLMNVDFADVRTVMSEMGYAMMGSGVASGEDRAEEAA
EMAISSPLLEDIDLSGARGVLVNITAGFDLRLDEFETVGNTIRAFASDNATVVIGTSLDP
DMNDELRVTVVATGIGMDKRPEITLVTNKQVQQPVLDRYQQHGMAPLTQEQKTVAKVVND
NAPQAAKEPDYLDIPAFLRKQAD - Wejdź na stronę BLAST na NCBI
- Wybierz "protein blast"
- Wklej poszukiwaną sekwencję
- W polu "Organism" wybierz bakterie i kliknij BLAST
- Na liście wyników możesz sprawdzić u jakich bakterii system odnalazł duże podobieństwo sekwencyjne
What other genes encode proteins that exhibit structures or motifs such as ones that have just been determined?
- Wejściowa struktura to łańcuch A z białka 1A1A
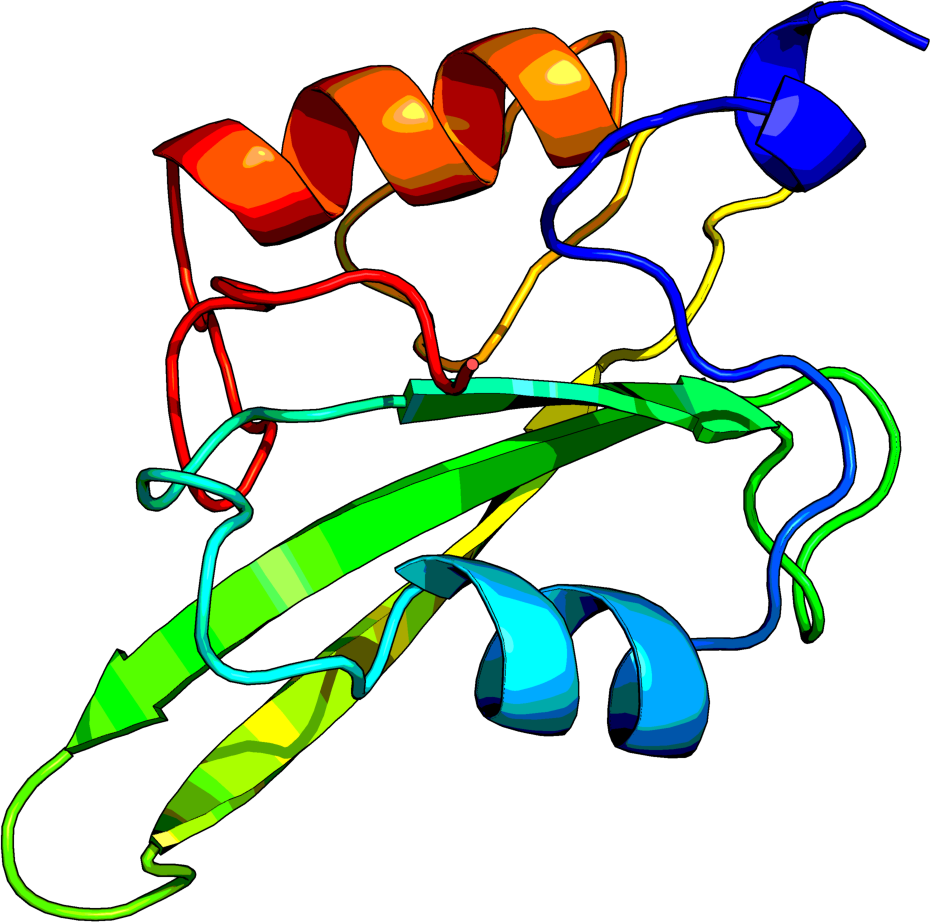
- FASTA:
>1a1a_A; molId:1; molType:protein; unp:P12931;
MDSIQAEEWYFGKITRRESERLLLNAENPRGTFLVRESETTKGAYSLSVSDFDNAKGLNV
KHYKIRKLDSGGFYITSRTQFNSLQQLVAYYSKHADGLCHRLTTVCP - Wyszukaj sekwencji w BLAST/BLAT (opcje: Homo sapiens, protein database)
- Na liście wyników, przejdź do strony z informacjami o genie (poprzez link w kolumnie "Overlapping Genes")
- Zobacz wyniki dla "Paralogues" (panel po lewej)
Informacje o strukturze przestrzennej
- Format danych PDB
- Plik tekstowy, składający się z różnych bloków, przede wszystkim nagłówka (komentarze, metadane, itp.) oraz współrzędnych atomów:
ATOM 1388 C5 C B 216 -51.777 0.122 2.841 1.00 0.00 C ATOM 1389 C6 C B 216 -52.775 0.660 3.557 1.00 0.00 C ATOM 1390 H5' C B 216 -53.536 1.485 7.588 1.00 0.00 H
- Baza danych: PDBe (europejska), PDB (główny, amerykański serwer), PDBj (japońska)
- Przejrzyj informacje odnośnie struktury 1EHZ
- Zwróć uwagę na:
- Tabelkę w sekcji Details
- Sekwencję nukleotydów w sekcji Structure / Primary
- Dane taksonomiczne odnośnie organizmu, z którego pochodzi RNA (wraz z odniesieniami)
- Informacje o publikacji w sekcji Citation
- Ligandy oraz reszty modyfikowane w Ligands
- A teraz sprawdź białko 1A1A
- W sekcji Structure / Primary nałożone są dodatkowo odnośniki do UniProt oraz Pfam (patrz listę innych baz danych poniżej)
- W sekcji Structure / Secondary można podejrzeć jaką formę przyjmują określone aminokwasy w strukturze
Wykorzystaj narzędzie Sequence search wpisując sekwencję z drugiego dzisiejszego zadania. Sprawdź znalezione struktury przestrzenne (linki "View" z kolumny PDB code).
Zadania programistyczne
Serwer PDB udostępnia interfejs programistyczny typu RESTful tzn. poprzez odwołania HTTP do odpowiednich adresów. Na stronie są też przykłady w Perlu, Pythonie i Javie.
Zadanie do wykonania brzmi następująco:
- Wejdź na stronę Advanced Search
- Ustal kryterium na: Release Date pomiędzy 2000-01-01 a 2000-12-31
- Dodaj kryterium: Macromolecular Type, zmień Contains RNA na yes
- Wykonaj zapytanie
- Na stronie wynikowej, kliknij w Query Details, zobaczysz swoje zapytanie w formacie XML; usuń z niego niepotrzebne wpisy: queryId, version, description, resultCount, runtimeStart, runtimeMilliseconds;
- Oczyszczony XML zapisz sobie jako szablon; przeanalizuj w jaki sposób zawiera on wpisane w formularzu dane: daty od/do oraz że interesują nas struktury zawierające RNA
- Napisz program, który:
- Otrzymuje na standardowym wejściu: datę "od", datę "do" oraz słowo kluczowe "Protein", "RNA", "DNA" lub "Hybrid"
- Modyfikuje odpowiednio szablon XML z zapytaniem
- Wywołuje zapytanie REST-owe na adres http://www.rcsb.org/pdb/rest/search/
- Zwraca użytkownikowi listę wyników
UWAGA! Jeśli podane zostanie słowo kluczowe np. "Protein" to dla pozostałych typów cząsteczek proszę podać w zapytaniu znak '?'.
Weryfikacja
1DQ7
1F8I
1F8M
$ ./program 2014-07-24 2014-07-31 RNA
4PMW
4PY5
4U1U
4U1V
4U20
4U24
4U25
4U26
4U27
4UJC
4UJD
Wykorzystaj inne udostępnione funkcje, żeby stworzyć program, który:
- Ze standardowego wejścia odczyta identyfikator PDB oraz nazwę łańcucha
- Wypisze opis wskazanego łańcucha (polymerDescription przy zapytaniu describeMol)
Weryfikacja
TRANSFER RNA (PHE)
$ ./program 1A1A A
C-SRC TYROSINE KINASE
$ ./program 100D A
DNA/RNA (5'-R(*CP*)-D(*CP*GP*GP*CP*GP*CP*CP*GP*)-R(*G)-3')
Inne bazy danych
- RNA FRABASE: an engine with database to search the three-dimensional fragments within 3D RNA structures
- NDB: contains information about experimentally-determined nucleic acids and complex assemblies
- BPS: a database of RNA base pairs with quantitative information on the spatial arrangements of interacting bases
- Modomics: presents RNA modification pathways on the level of nucleosides
- GenBank: an annotated collection of all publicly available DNA sequences
- BioModels: a reliable repository of computational models of biological processes
- Pfam, Rfam: a large collection of protein/RNA families
- UniProt: a comprehensive, high-quality and freely accessible resource of protein sequence and functional information
- SwissProt: an annotated and non-redundant protein sequence database