Motywy strukturalne
- Stem
(((())))
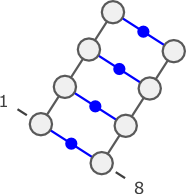
- Hairpin
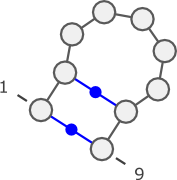
((.....))
- Bulge
(((((((())))..))))
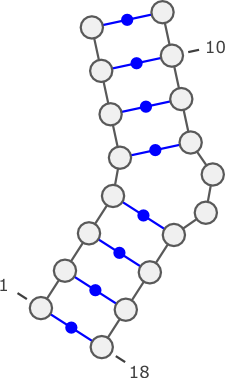
- Internal loop
((((...(((())))..))))
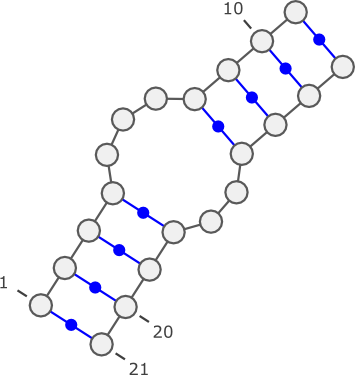
- 3-way junction
((((...((())).....((()))..))))
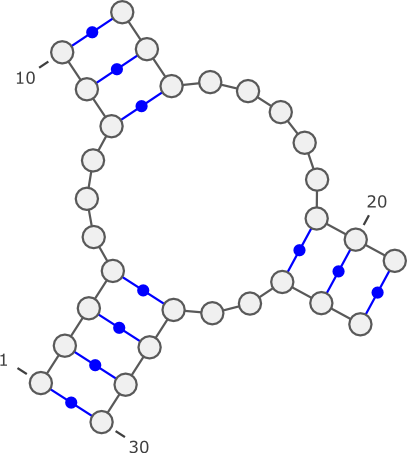
- 4-way junction
((((...((())).....((()))..(()).........))))
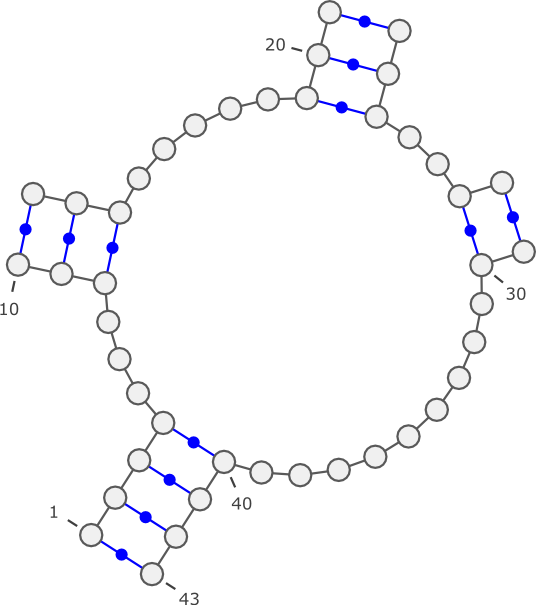
- Pseudoknot
...[[..((((..]]....))))..
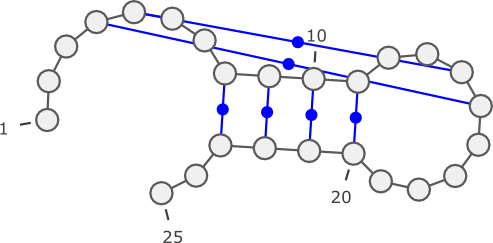
- Kissing loops
((((..[[..))))((((..]]..))))
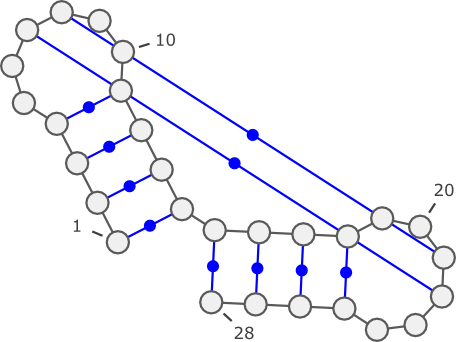
Wizualizacja struktury drugorzędowej
Wykonaj kroki w poniższych sekcjach. Otrzymane wyniki tekstowe, grafiki (np. zrzuty ekranu z okienkiem) i/lub swoje komentarze umieść w krótkim raporcie/sprawozdaniu.
VARNA
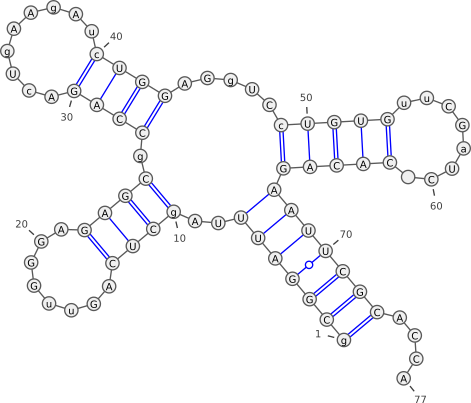
- Uruchom VARNA, zobaczysz dwie wczytane domyślnie struktury
- Zapisz strukturę "User file #2" we wszystkich wspieranych formatach (prawy przycisk myszy → Save as)
- Spróbuj "chwycić" i przeciągnąć fragment dwuniciowy
- Zaznacz dowolny fragment struktury (kliknij w dowolnym miejscu i trzymając przycisk myszy zaznaczaj... tak jak ikony na pulpicie :)). Zwróć uwagę jak to jest pokazane w polu z sekwencją i strukturą
- Dodaj wszystkie automatyczne adnotacje (prawy przycisk myszy → Annotations → Auto...)
- Zaznacz wizualnie jedno z ramion pętli I2 zawierające: H3, I4, H4 oraz T1 (prawy przycisk myszy → Annotations → New → Region)
- Pokaż strukturę we wszystkich dostępnych metodach rysowania (prawy przycisk myszy → Redraw → Algorithm → ...)
- A teraz dodaj parowania między pętlą T1 (nukleotydy AIA) oraz T2 (nukleotydy IAC). W tym celu zmień wartość w polu ze strukturą drugorzędową z kropek na odpowiednie nawiasy zamykające i otwierające. Jakie nawiasy będą odpowiednie i dlaczego?
- Ponownie narysuj strukturę we wszystkich czterech sposobach wizualizacji. Jak dodanie trzech par z poprzedniego punktu wpłynęło na wynik, w szczególności dla algorytmu Linear oraz Circular?
PseudoViewer
- Wejdź na stronę z formularzem PseudoViewer
- Narysuj motywy Pseudoknot oraz Kissing loops przedstawione wyżej na stronie (skopiuj dot-bracket do pola Structure; sekwencja jest nieistotna w tym ćwiczeniu, możesz wpisać jedną literę
Aw polu Sequence, to wystarczy) - Zastąp pseudowęzły niesparowanymi resztami (w miejsce nawiasów kwadratowych wpisz kropki) i wygeneruj grafiki ponownie
- Spróbuj także z poniższą większą strukturą i porównaj wyniki z grafikami rysowanymi przez VARNA:
CAGCACGACACUAGCAGUCAGUGUCAGACUGCAIACAGCACGACACU
AGCAGUCAGUGUCAGACUGCAIACAGCACGACACUAGCAGUCAGUGU
CAGACUGCAIA
.[(((((...(((((...(((((........(((((...(((((..]
..)))))...)))))..................))))).....))))
)...))))).. - Zasymuluj "braki" w strukturze. Przełącz typ wejścia na PseudoViewer input type. Jest to format kropkowo-nawiasowy, w którym można przeplatać na zmianę pary linii sekwencja-struktura. Jeśli linie poprzedzone są numerem, to na wygenerowanej grafice nukleotydy mają nowe numerowanie. Sprawdź przykład:
1 CCUUACCUCGGGUAGAGGCCCA
1 (((......)))..(())....
120 CCUUACCUCGGGUAGAGGCCCA
120 (((......)))..(())....
forna
- Wyświetl wizualizację struktury z VARNA (User file #2) przy użyciu programu forna.
Uwaga! Wymagana jest zamiana literyIw sekwencji na inną np.A - Wypróbuj różne opcje kolorowania i wyświetlania
- Wyświetl również wizualizację struktury kompleksu RNA-białko np. 4TZP
Przewidywanie struktury drugorzędowej na podstawie sekwencji
- Jeden z pierwszych powszechnie wykorzystywanych algorytmów do przewidywania struktury drugorzędowej zaproponowała Ruth Nussinov (link do publikacji z 1980 roku)
- Opierał się on na metodzie programowania dynamicznego
- Dla zadanej sekwencji, algorytm odnajduje maksymalną liczbę parowań A-U oraz G-C
- Algorytmów do przewidywania struktur drugorzędowych jest obecnie bardzo dużo. Przykładowe to:
- RNAstructure - jest to cały pakiet programów o nieco różnym przeznaczeniu np. ProbKnot wyznacza progi prawdopodobieństwa parowania się dwóch nukleotydów wliczając w to interakcje typu pseudowęzła
- mfold - działa w oparciu o reguły termodynamiczne odnajdując struktury o minimalnej energii swobodnej
- Warto też sprawdzić stronę CompaRNA, która zawiera rankingi metod do przewidywania struktur drugorzędowych
- CompaRNA opiera się na liście znanych struktur drugorzędowych i na automatycznym uruchamianiu wszystkich wspieranych metod
- Wyniki są następnie porównywane parami ze strukturą referencyjną i oceniane jest ich podobieństwo
- Oczywiście większość metod do przewidywania struktur posiada zestawy parametrów do ustalenia, a CompaRNA działa tylko na domyślnych wartościach. Mimo to CompaRNA oferuje bardzo przydatny i możliwie obiektywny (bo automatyczny) ranking metod do przewidywania struktur drugorzędowych
Przetestuj dwa wskazane wcześniej narzędzia do przewidywania struktury drugorzędowej oraz jedno wybrane przez Ciebie z listy na stronie CompaRNA. Porównaj zwracane przez nie wyniki.
Sekwencję do testów pobierz tutaj
Uwaga! Sekwencja generowana jest losowo przy każdym odświeżeniu strony, więc należy ją sobie zapisać gdzieś osobno!
Napisz program, który przetworzy plik wejściowy w formacie BPSEQ i wypisze wszystkie motywy typu stem. Każdy motyw ma być opisany w jednej linii jako para liczb - indeksów początku i końca licząc od strony 5'.
Przykład: (1EHZ-2D-bpseq.txt)
$ ./program.py input.bpseq
1 7
10 13
27 30
49 53
1 7
10 13
27 30
49 53