Wstęp
Pętle typu GNRA
- Pętla typu GNRA (gdzie
Nto dowolny nukleotyd, czyliA,C,GlubU, aRto puryna czyliAlubG) to najczęstszy motyw strukturalny RNA - Cechą charakterystyczną wszystkich 8 możliwych układów jest niekanoniczne wiązanie G-A stabilizujące układ
- Dwa pozostałe nukleotydy
NiRtworzą wiązania wodorowe z odległymi fragmentami jednoniciowymi lub białkami - Utrzymują się w ten sposób przestrzenne interakcje wpływające na całościowe zwinięcie się struktury
- Biorą one udział w stabilizacji, szczególnie dla dużych struktur RNA (1000+ nukleotydów)
- Wizualizacja poniżej przedstawia strukturę drugorzędową dla 1ZIH (tutaj pętla to GCAA):
Symulacje dynamiki molekularnej
- Symulacja dynamiki molekularnej to iteracyjne powtarzanie rozwiązywania równań ruchu Newtona dla układu atomów (powiązanych odpowiednimi ograniczeniami wynikającymi ze struktury i właściwości własnych atomów)
- Punktem początkowym jest struktura (współrzędne wszystkich jej atomów), najczęściej umieszczona w wodzie z jonami celem zobojętnienia
- Układ posiada też temperaturę (czyli de facto wektory prędkości dla każdego z atomów) i znajduje się pod określonym ciśnieniem
- Każdy krok symulacji odpowiada niewielkiej zmianie czasowej (często 1 fs, czyli 1e-15 s)
- Symuluje się różnej długości trajektorie w zależności od problemu badawczego
- Dynamika molekularna pozwala też na:
- sprawdzenie stabilności struktury po wprowadzeniu modyfikacji lub mutacji,
- analizę interakcji czwartorzędowych (np. przeprowadzenie tzw. dokowania ligandu do miejsca aktywnego białka),
- pełnoatomowe potwierdzenie eksperymentalnych wyników (np. temperatura topnienia RNA/DNA to temperatura, w której 50% składu próbki ulega denaturacji; przy pomocy symulacji można ten proces prześledzić dokładniej),
- przewidzenie struktury trzeciorzędowej na podstawie jej sekwencji,
- i wiele innych…
- Poniższy film przedstawia symulację pętli GNRA, w której autorom udało się z formy w pełni rozwiniętej (unfolded) otrzymać końcową postać znaną z wielu innych badań strukturalnych: (źródło: High-Resolution Reversible Folding of Hyperstable RNA Tetraloops Using Molecular Dynamics Simulations)
Przeprowadzenie własnej symulacj
Wymagane oprogramowanie
Pliki potrzebne do przeprowadzenia symulacji
Początkowa struktura (współrzędne atomów) w formacie PDB (w naszym przypadku: 1ZIH
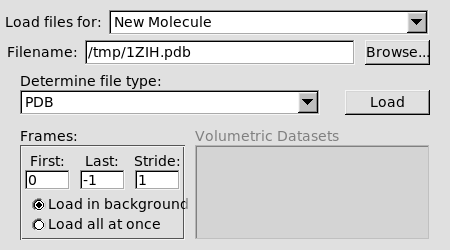
- W okienku VMD Main wybieramy menu File → New Molecule
- Poprzez Browse wybieramy plik
1ZIH.pdbi klikamy Load
Informacja o strukturze w formacie PSF:
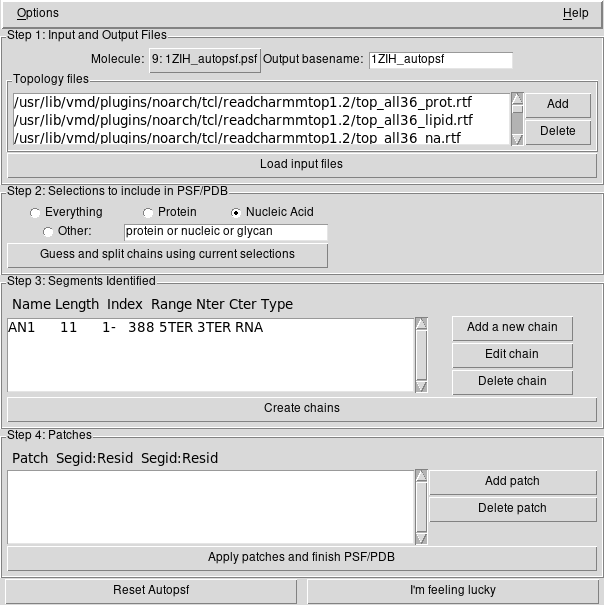
- W okienku VMD Main wybieramy menu Extension → Modeling → Automatic PSF Builder
- W okienku AutoPSF w menu Options zaznaczamy Add solvation box i Add neutralizing ions
- W sekcji Step 1: … klikamy Load input files
- W sekcji Step 2: … zaznaczamy Nucleic Acid i klikamy Guess and split …
- W sekcji Step 3: … klikamy Create chains
Symulacja przy pomocy AutoIMD (NAMD)
W okienku VMD Main wybieramy menu Extensions → Simulations → AutoIMD (NAMD)
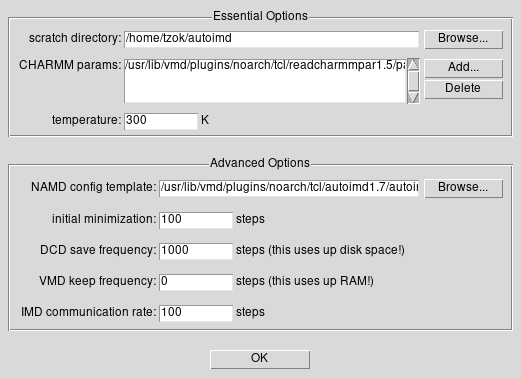
W okienku AutoIMD Controls wybieramy menu Settings -> Simulation Parameters
- Zmieniamy DCD save frequency na 1000 (jest to częstotliwość zapisu “klatek” w trajektorii)
- Zmieniamy IMD communication rate na 100 (jest to częstotliwość aktualizowania VMD)
W okienku AutoIMD Controls wybieramy menu File → Show Simulation Window
W okienku AutoIMD Controls w polu imdmolten (ruchome atomy) wpisujemy nucleic i klikamy Submit
- Kliknięcie Connect uruchomi symulację, ale zanim to się stanie polecam zmienić sposób wizualizacji struktury RNA:
- W okienki VMD Main mamy listę struktur i plików PSF, dwukrotnie klikamy na litery
Dwe wszystich wierszach oprócz AutoIMD Simulation. Dwukrotne kliknięcie zmieni literęDna czerwoną i sprawi, że dane atomy nie będą widoczne, zatem przestaniemy widzieć atomy tworzące wodę i jony - W okienku VMD Main wybieramy menu Graphics → Representations
- Ustawiamy Selected Molecule na AutoIMD Simulation
- Klikamy Create Rep
- W polu Selected Atoms wpisujemy nucleic
- Jako Drawing Method wybieramy HBonds
- Jako Coloring Method wybieramy ColorID z wartością 4
- Możemy też zwiększyć Line Thickness do wartości 4
- W głównym oknie VMD widzimy teraz tylko RNA oraz na żółto zaznaczone wiązania wodorowe między zasadami
- W okienki VMD Main mamy listę struktur i plików PSF, dwukrotnie klikamy na litery
- Gdy w okienku AutoIMD Controls klikniemy Connect to uruchomi się symulacja, domyślnie etap equilibration. Można oglądać efekty w głównym oknie VMD oraz w logach w okienku IMD Connection
- Etap ten kontynuuje się dopóki zachodzą jakieś znaczące zmiany, ale w momencie gdy system zaczyna oscylować wokół tych samych stanów można zatrzymać symulację klikając Pause (na potrzeby eksperymentu, zatrzymajmy symulację po 10 tys. kroków = 20 ps czasu)
- Tryb symulacji zmieniamy w okienku AutoIMD Controls w menu Settings → Minimization Mode i uruchamiamy ciąg dalszy klikając Pause. Minimalizację przeprowadza się dopóki symulacja nie zatrzyma się w optimum lokalnym (na potrzeby eksperymentu, zatrzymajmy symulację po 40 tys. krokach)
Analiza przebiegu symulacji
Do wykonania kroków poniżej sugeruję zacząć z czystą sesją VMD:
- W okienku VMD Main zaznaczamy wszystkie wiersze i wybieramy z menu Molecule → Delete Molecule
- Ewentualnie można wyłączyć i włączyć ponownie VMD :)
Namd generuje zestaw plików wyjściowych, a plugin AutoIMD domyślnie umieszcza je w
$HOME/autoimd. Nas interesować będzieautoimd.log(tekstowy log z przebiegu symulacji) orazautoimd.dcd(binarnie zapisana trajektoria = pozycje atomów zapisywane co pewną liczbę iteracji)Aby otworzyć trajektorię w VMD:
- Wybieramy z menu File → New Molecule
- Upewnijmy się, że pole Load files for: ma wartość New Molecule
- Wybieramy plik
autoimd.psf(Uwaga! Lepiej wybrać plik PSF niż PDB. Dla tego drugiego wczytanie trajektorii też zadziała, ale wówczas VMD nie skorzysta z pełni informacji o strukturze jaka jest tylko w PSF) - Po naciśnięciu Load okienko wczytywania powinno zostać aktywne, a pole Load files for: zmieni wartość na
autoimd.psf - Wybieramy plik
autoimd.dcdi klikamy Load
Aby przeanalizować plik logu w VMD:
- Wybieramy z menu Extensions → Analysis → NAMD Plot
- W nowym okienku wybieramy File → Select NAMD Log File i wskazujemy na plik
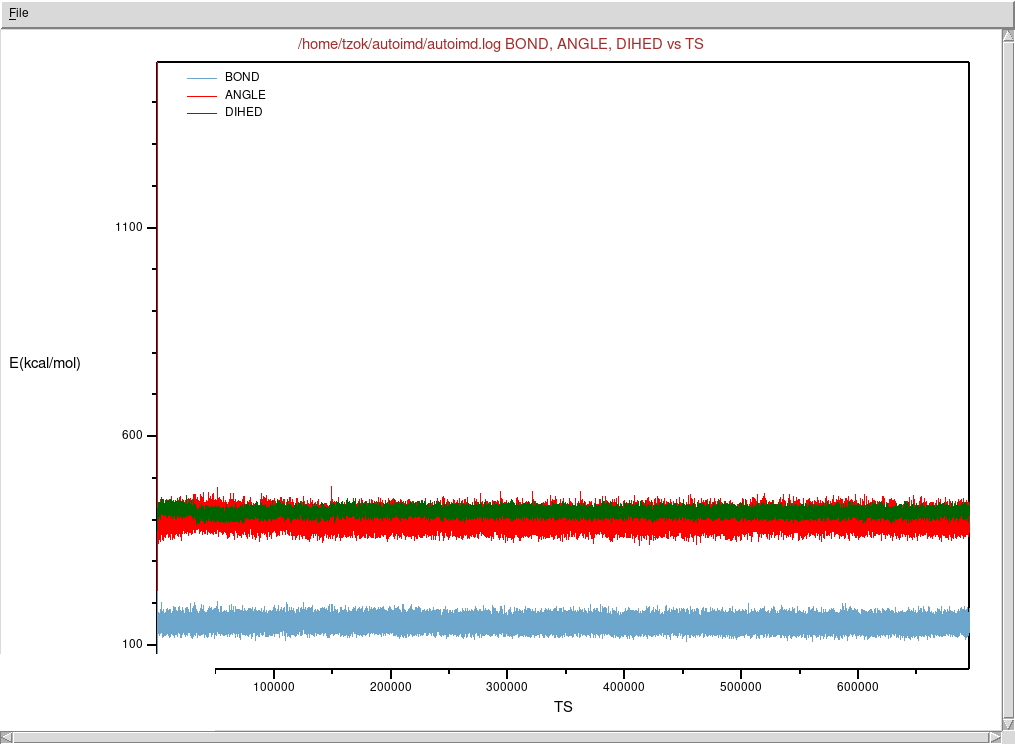
autoimd.log - Zaznaczmy przykładowo BOND, ANGLE oraz DIHED odpowiadające trzem komponentom funkcji energii w modelu
- Wybieramy z menu File → Plot Selected Data
Analiza gotowej, dłuższej trajektorii
Pełniejszą i trójetapową symulację (minimalizacja, podgrzewanie, swobodny ruch) możemy przeanalizować dzięki uprzejmości dr Joanny Sarzyńskiej
- Pobierzmy i rozpakujmy archiwum
- Wybieramy w VMD File → New Molecule
- Upewnijmy się, że Load files for: ma wartość New Molecule
- Wskazujemy na plik
1ZIH_js.psf(ponownie, nleży użyć pliku PSF, a nie PDB) i klikamy Load - We wciąż otwartym oknie Molecule File Browser, z polem Load files for: ustawionym na
1ZIH_js.psfwskazujemy na plikmin.dcdz pobranego archiwum i klikamy Load - Powtaramy poprzedni krok dla
heat.dcdi następnierun1.dcd(w taki właśnie sposób “doczytuje” się kolejne ramki z zapisanej trajektorii, VMD dokłada je na końcu tworząc spójny, jednolity obraz symulacji)
Uruchom całą symulację (przycisk ▶ w oknie VMD Main), możesz sterować suwakiem speed by spowolnić wizualizację kolejnych kroków
Sprawdźmy przebieg zmian strukturalnych w czasie trwania symulacji:
- Wybieramy z menu głównego okna VMD Extension → Analysis → RMSD Trajectory Tool
- W polu edycyjnym w lewym górnym rogu (jest to zaznaczenie fragmentu, którego ma dotyczyć analiza) wpisujemy nucleic
- Klikamy najpierw przycisk ALIGN, następnie RMSD
- Wybieramy w menu File → Plot Data aby zobaczyć wykres zmieniającego się RMSD w zależności od numeru ramki (Uwaga! Różne etapy symulacji miały różny odstęp czasowy pomiędzy zapisem współrzędnych do pliku trajektorii. Oznacza to, że między sąsiednimi ramkami nie zawsze jest ta sama różnica w czasie)
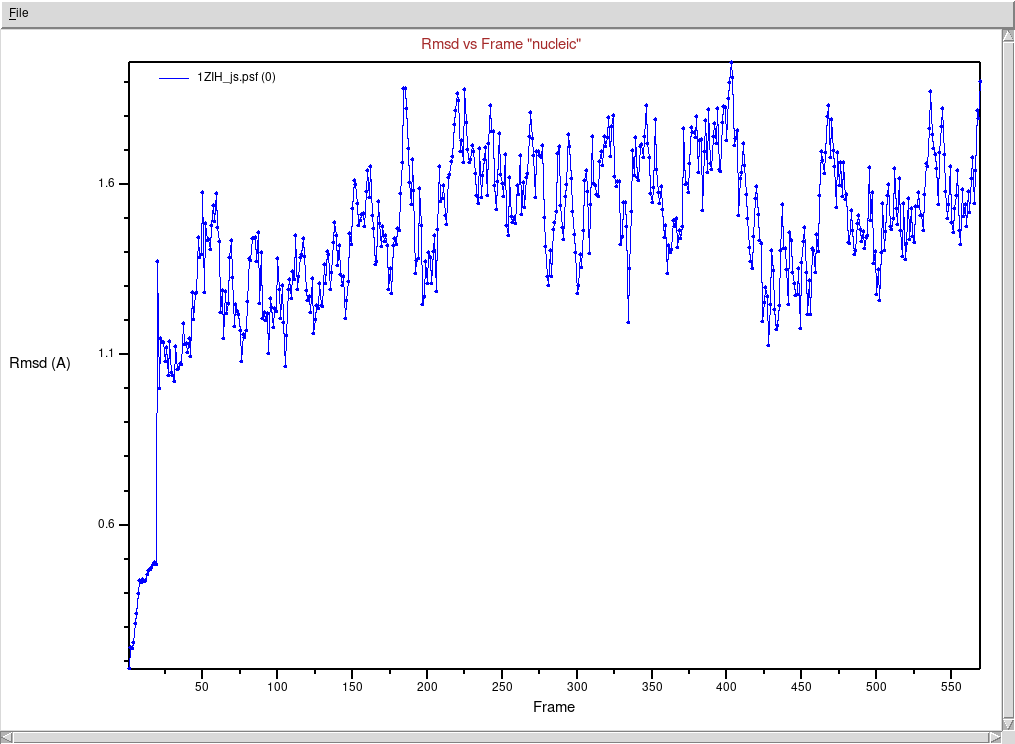
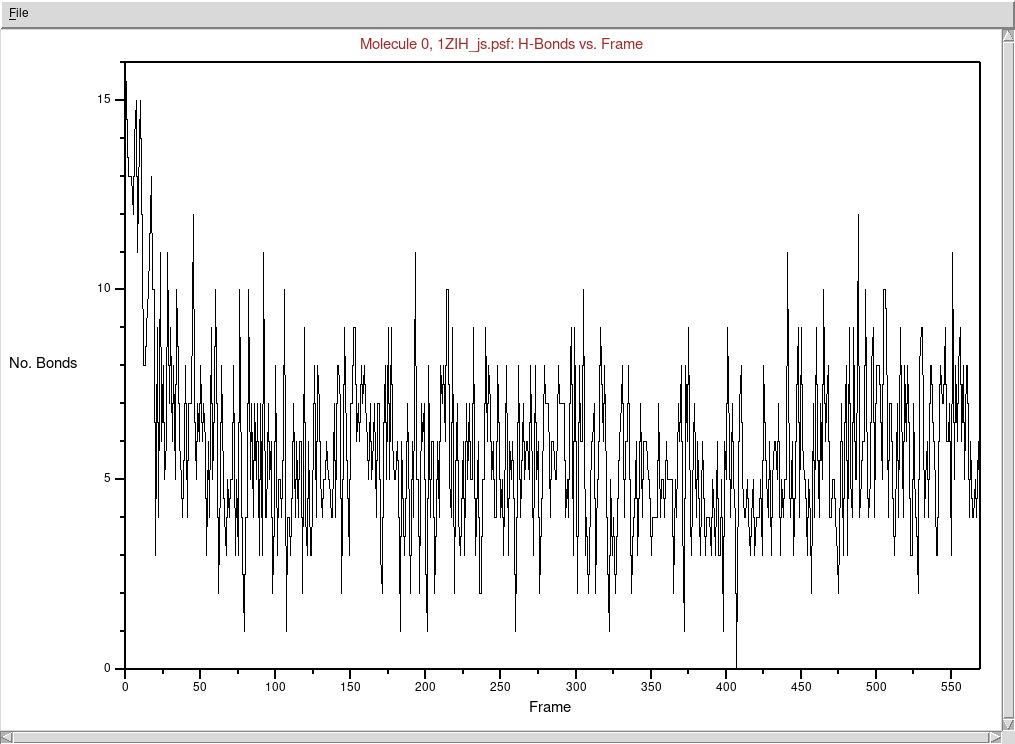
Sprawdźmy zmiany liczby wiązań wodorowych w czasie trwania symulacji:
- Wybieramy z menu głównego okna VMD Extensions → Analysis → Hydrogen Bonds
- Jako Selection 1 podajemy nucleic
- Klikamy Find hydrogen bonds